# 1. Cargar paquetes necesarios
library(osfr)
library(tidyverse)
library(ergm)
library(Matrix)
library(network)
library(ggraph)
library(tidygraph)
library(kableExtra)
library(igraph)
library(sna)
library(plotly)
library(plotrix)
library(texreg)
# Configurar tema para visualizaciones
theme_set(theme_minimal())Introducción
La ciencia de la sustentabilidad requiere urgentemente de innovación transformadora para abordar desafíos socio-ambientales complejos. Comprender cómo emerge la investigación disruptiva en las redes científicas es, por tanto, crucial. Li, et. al. (2024) han demostrado una relación inversa entre productividad y disrupción científica, pero los mecanismos estructurales subyacentes a este fenómeno permanecen poco claros, particularmente en el contexto de redes modo-2 que vinculan autores y publicaciones.
La sociología del conocimiento científico ha identificado históricamente la dualidad fundamental entre las estructuras sociales de producción y el contenido del conocimiento producido (Mannheim 1936; Durkheim [1893] 1933). Esta dualidad se manifiesta particularmente en campos emergentes como la ciencia de la sustentabilidad, donde la innovación conceptual y la estructura social de producción se constituyen mutuamente. A diferencia de visiones que enfatizan la fragmentación disciplinar o la mera acumulación de capital científico, proponemos que la producción de conocimiento científico emerge de la interacción entre mecanismos de cohesión estructural, procesos de legitimación y dinámicas de innovación disruptiva.
El trabajo seminal de Gondal (2011) sobre campos emergentes identifica cómo la incertidumbre inicial y la necesidad de legitimación producen una centralización temprana que estructura el desarrollo posterior del campo. Sin embargo, esta centralización no es simplemente un proceso de acumulación de capital científico como sugería Bourdieu (1975), sino que refleja una necesidad estructural más profunda: establecer las bases sociales compartidas que permiten la evaluación y validación del conocimiento en condiciones de alta incertidumbre. Esto se vincula con lo que Kuhn (1970) denominaba “ciencia normal” - la necesidad de establecer paradigmas que permitan el trabajo científico acumulativo. Sin embargo, mientras Kuhn veía los paradigmas principalmente como estructuras cognitivas, el análisis de redes modo-2 revela que estos se sostienen en estructuras relacionales específicas que emergen de patrones sistemáticos de colaboración. Moody (2004) profundiza esta comprensión al demostrar que la cohesión estructural que emerge en las redes de colaboración no es un simple subproducto de la centralización, sino un mecanismo generativo fundamental. Esta cohesión estructural permite la integración del conocimiento incluso en condiciones de alta especialización a través de lo que Friedkin (1998) denominó “overlapping consensus” - consensos parciales superpuestos que facilitan la comunicación entre diferentes tradiciones especializadas.
Los hallazgos de Wu et al. (2019) sobre equipos pequeños y disrupción científica revelan una tensión productiva inherente a los campos científicos: mientras la cohesión estructural facilita la acumulación y validación del conocimiento, la innovación disruptiva requiere posiciones estructurales que permitan cierta autonomía respecto a los paradigmas dominantes. Esta tensión no es una simple contradicción, sino un principio organizador fundamental de los campos científicos que se resuelve a través de mecanismos de diferenciación integrada.
En el caso específico de la ciencia de la sustentabilidad, la urgencia transformadora modifica la evolución típica de los campos científicos. La necesidad apremiante de innovación para abordar desafíos socio-ambientales complejos genera una mayor tolerancia temprana a innovaciones disruptivas y promueve estructuras más distribuidas de producción de conocimiento. Esto resulta particularmente evidente en la relación inversa entre productividad y disrupción identificada por Li et al. (2024), donde los actores centrales tienden a desarrollar líneas establecidas mientras que la innovación disruptiva emerge frecuentemente desde posiciones periféricas.
La dimensión temporal es crucial para entender cómo estos mecanismos operan en diferentes etapas del desarrollo del campo. Los campos científicos emergentes atraviesan fases de legitimación inicial, consolidación y diferenciación, y eventual maduración e integración dinámica. En cada fase, la relación entre posición estructural y capacidad disruptiva evoluciona de manera diferenciada, mostrando inicialmente una fuerte centralización para establecer legitimidad, seguida de una diversificación de las fuentes de innovación y eventualmente alcanzando un equilibrio dinámico entre desarrollo y disrupción.
Mecanismos Generativos y Propiedades Emergentes
Los hallazgos de Wu et al. (2019) sobre equipos pequeños y disrupción científica revelan una tensión productiva inherente a los campos científicos: mientras la cohesión estructural facilita la acumulación y validación del conocimiento (Moody 2004), la innovación disruptiva requiere posiciones estructurales que permitan cierta autonomía respecto a los paradigmas dominantes. Esta tensión no es una simple contradicción, sino un principio organizador fundamental de los campos científicos.
Esta dinámica se resuelve a través de lo que denominamos “mecanismos de diferenciación integrada”:
La cohesión estructural (Moody 2004):
- Provee el sustrato necesario para la comunicación y validación del conocimiento
- Facilita la difusión de estándares metodológicos y criterios de evaluación
- Permite la especialización sin fragmentación total
- Opera a través de patrones sistemáticos de colaboración que crean puentes entre diferentes áreas
La centralización temprana (Gondal 2011):
- Establece estándares y criterios de evaluación compartidos
- Reduce la incertidumbre inicial sobre qué constituye conocimiento válido
- Crea puntos de referencia comunes para el desarrollo del campo
- Facilita la acumulación inicial de conocimiento validado
La autonomía estructural (Wu, Wang, and Evans 2019):
- Permite la exploración de nuevas direcciones teóricas y metodológicas
- Se asocia con posiciones periféricas o semi-periféricas en la red
- Facilita el cuestionamiento de supuestos establecidos
- Opera principalmente a través de equipos pequeños con mayor libertad para innovar
La Diferenciación como Proceso Relacional
La diferenciación entre campos STEM y SHAPE emerge no tanto de divisiones epistemológicas fundamentales sino de diferentes regímenes de validación del conocimiento que se cristalizan en patrones distintivos de vinculación autor-artículo. Estos regímenes reflejan diferentes:
- Criterios de validación empírica
- Estructuras de argumentación aceptadas
- Formas legítimas de construcción teórica
- Patrones típicos de colaboración
- Mecanismos de acreditación y reconocimiento
Temporalidad y Evolución en Campos Científicos Emergentes
La dimensión temporal es crucial para entender cómo los mecanismos generativos operan en diferentes etapas del desarrollo de campos emergentes. Siguiendo a Gondal (2011), la evolución de estos campos no es lineal sino que exhibe tensiones dinámicas entre legitimación, innovación y estructuración.
Fases de Desarrollo Estructural
- Fase de Emergencia y Legitimación Inicial:
- Alta incertidumbre sobre criterios de validación
- Centralización temprana como mecanismo de reducción de incertidumbre
- Dependencia de autoridades externas para legitimación
- Formación inicial de grupos pequeños altamente cohesivos
- Fase de Consolidación y Diferenciación:
- Desarrollo de criterios internos de validación
- Emergencia de estructuras centro-periferia
- Diversificación de líneas de investigación
- Formación de subcampos especializados mientras se mantiene cohesión general
- Fase de Maduración e Integración Dinámica:
- Equilibrio entre innovación disruptiva y desarrollo acumulativo
- Estructuras de colaboración más complejas y multinivel
- Mecanismos establecidos para integrar conocimiento diverso
- Capacidad dual para mantener coherencia y generar disrupciones
El Caso de la Ciencia de la Sustentabilidad
La ciencia de la sustentabilidad representa un caso particularmente relevante de campo científico emergente por varias razones:
- Urgencia Transformadora:
- Necesidad apremiante de innovación para abordar desafíos socio-ambientales complejos
- Tensión intrínseca entre la urgencia de soluciones y la necesidad de rigor científico
- Demanda de disrupciones que transformen paradigmas establecidos
- Complejidad Estructural:
- Naturaleza inherentemente interdisciplinaria
- Necesidad de integrar ciencias naturales y sociales
- Múltiples comunidades epistémicas con diferentes tradiciones
- Dinámica Centro-Periferia Específica:
- Li et al. (2024) identifican una relación inversa entre productividad y disrupción
- Los actores centrales tienden a desarrollar líneas establecidas
- La innovación disruptiva emerge frecuentemente desde posiciones periféricas
Implicaciones para la Estructura de Colaboración
Esta conceptualización temporal tiene importantes implicaciones para entender cómo evolucionan las estructuras de colaboración:
- Evolución de Patrones Colaborativos:
- De colaboraciones principalmente diádicas a estructuras más complejas
- Desarrollo de “brokers” que conectan diferentes comunidades epistémicas
- Emergencia de equipos especializados en diferentes tipos de contribución
- Transformación de Mecanismos de Legitimación:
- De dependencia externa a criterios internos de validación
- Desarrollo de estándares específicos del campo
- Evolución de mecanismos de evaluación que balancean rigor e innovación
- Dinámica de Innovación:
- as fuentes de innovación se diversifican con el tiempo
- os roles estructurales (central vs. periférico) se vuelven más fluidos
- merge una división del trabajo entre desarrollo y disrupción
Esta perspectiva temporal enriquece nuestra comprensión de cómo los mecanismos generativos identificados anteriormente operan y evolucionan a lo largo del tiempo, sugiriendo que la capacidad de un campo para mantener su vitalidad depende de su habilidad para institucionalizar tensiones productivas entre diferentes modos de producción de conocimiento.
Hipótesis
H1: Estructura Base y Centralización
“La red mostrará una baja densidad base pero con patrones de centralización asimétricos entre papers y autores.”
Justificación: Gondal (2011) argumenta que los campos emergentes muestran inicialmente baja densidad debido a la incertidumbre estructural, pero desarrollan patrones de centralización que reflejan la necesidad de establecer legitimidad. Moody (2004) complementa esta visión señalando que diferentes tipos de actores (en nuestro caso, papers y autores) desarrollan roles estructurales distintos en el proceso de legitimación del campo.
H2: Asimetría Disciplinar
“Los patrones de colaboración entre campos mostrarán asimetrías direccionales, con diferentes probabilidades de conexión según el campo de origen del paper y el campo del autor.”
Justificación: Friedkin (1998) sugiere que la integración del conocimiento ocurre a través de “consensos superpuestos” que permiten la comunicación entre tradiciones especializadas. Esta integración, según Moody (2004), no es simétrica sino que refleja diferentes regímenes de validación del conocimiento entre campos.
H3: Dinámica Centro-Periferia y Disrupción
“La capacidad disruptiva mostrará una relación inversa con la centralidad estructural, moderada por el campo disciplinar.”
Justificación: Li et al. (2024) identifican una relación inversa entre productividad y disrupción, donde los actores centrales tienden a desarrollar líneas establecidas mientras que la innovación disruptiva emerge desde posiciones periféricas. Wu et al. (2019) complementan esta visión mostrando cómo los equipos pequeños, típicamente más periféricos, son más propensos a la disrupción.
H4: Evolución Temporal y Campos Emergentes
“La relación entre posición estructural y capacidad disruptiva evolucionará de manera diferenciada según la fase de desarrollo del campo, con patrones distintos entre STEM y SHAPE.”
Justificación: Gondal (2011) describe cómo los campos emergentes evolucionan a través de fases de legitimación inicial, consolidación y diferenciación. En campos como la ciencia de la sustentabilidad, esta evolución se ve modificada por la urgencia transformadora, produciendo:
- Mayor tolerancia temprana a innovaciones disruptivas
- Estructuras más distribuidas de producción de conocimiento
- Mecanismos acelerados de validación y difusión
H5: Integración Disciplinar Asimétrica
“Los campos mostrarán diferentes capacidades de integración, con STEM actuando como puente entre tradiciones disciplinares.”
Justificación: La sociología del conocimiento científico (e.g., Mannheim 1936; Kuhn 1970) sugiere que diferentes campos desarrollan distintas capacidades de integración basadas en sus tradiciones epistemológicas. Bourdieu (1975) complementa esta visión señalando cómo algunos campos acumulan capital científico que les permite actuar como integradores.
Metodología
La complejidad teórica identificada en la producción de conocimiento en campos científicos emergentes requiere una estrategia metodológica que integre diferentes aproximaciones analíticas. Proponemos combinar el análisis de cohesión estructural desarrollado por Moody (2004) con modelos exponenciales de grafos aleatorios (ERGMs) modo-2 siguiendo a Gondal (2011), para examinar cómo la dualidad entre productores y productos del conocimiento genera patrones específicos de colaboración y disrupción.
Cohesión Estructural y Patrones de Colaboración
Moody (2004) demuestra que el análisis de bicomponentes en redes de coautoría proporciona una medida robusta de cohesión estructural que permite identificar cómo la especialización disciplinar coexiste con la integración del conocimiento. Un bicomponente, al requerir múltiples caminos independientes entre cada par de nodos, identifica grupos que mantienen su conectividad incluso ante la remoción de miembros individuales. Esta propiedad es particularmente relevante para entender la robustez de las estructuras de colaboración científica. Siguiendo esta aproximación, analizaremos primero la red unimodal de coautoría para:
- Identificar la evolución del componente gigante y su relación con la fragmentación disciplinar
- Examinar la distribución y composición de bicomponentes como indicadores de cohesión
- Evaluar la robustez de las estructuras de colaboración a través del tiempo
Dualidad Estructural y ERGMs Modo-2
Complementando el análisis de cohesión, los ERGMs modo-2 desarrollados por Gondal (2011) permiten modelar explícitamente la dualidad entre autores y artículos que caracteriza la producción científica. Esta aproximación modela la probabilidad de observar un vínculo autor-artículo como función de:
- Características de los autores (productividad, centralidad previa)
- Atributos de los artículos (disrupción, impacto)
- Configuraciones estructurales locales (patrones de coautoría)
- Variables contextuales (campo disciplinar, período temporal)
Estrategia de Modelamiento Integrada
Nuestra estrategia procede en tres etapas:
- Análisis de Cohesión Estructural:
- Identificación de componentes y bicomponentes
- Evaluación de patrones de especialización vs integración
- Análisis de evolución temporal de estructuras cohesivas
- Modelamiento ERGM Modo-2:
- Especificación base con efectos de grado y homofilia
- Incorporación de términos estructurales complejos
- Modelamiento de interacciones entre disrupción y posición estructural
- Integración de Resultados:
- Vinculación entre patrones macro de cohesión y mecanismos generativos locales
- Evaluación de cómo diferentes configuraciones estructurales facilitan o inhiben la innovación disruptiva
- Análisis de evolución temporal de mecanismos relacionales
Operacionalización y Medición
Disrupción Científica: Siguiendo a Wu et al. (2019) y Li et al. (2019), medimos la disrupción a través de patrones de citación que indican en qué medida un artículo reorienta la atención del campo hacia nuevas direcciones.
Cohesión Estructural: Operacionalizada a través de:
- Tamaño y composición de bicomponentes
- Distribución de k-componentes
- Medidas de conectividad robusta
Posición Estructural: Capturada mediante:
- Centralidad en la red de coautoría
- Patrones de vinculación autor-artículo
- Pertenencia a componentes cohesivos
Validación y Control La validez del análisis se asegura mediante:
Controles por factores exógenos:
- Efectos temporales
- Diferencias disciplinares
- Variación institucional
Diagnósticos específicos:
- Convergencia de modelos ERGM
- Bondad de ajuste de configuraciones estructurales
- Robustez a diferentes especificaciones
Validación cruzada entre métodos:
- Consistencia entre patrones macro y micro
- Triangulación de resultados
- Evaluación de hipótesis alternativas
Esta estrategia metodológica integrada permite examinar cómo la dualidad estructural característica de la producción científica genera simultáneamente cohesión disciplinar e innovación disruptiva, proporcionando una comprensión más profunda de la evolución de campos científicos emergentes como la ciencia de la sustentabilidad.
Configuración Inicial y Carga de Datos
# 2. datos
load("/media/rober/4612-9FBE/recup_22112024/Documents/rcantillan.github.io/blog/posts/04-twomode-ergm/data/b3_fromlatam_1990_2000.RData")
#glimpse(b3_fromlatam_1990_2000)
# 3. Filtrar datos de Latam
#b3_fromlatam <- b3_joined %>%
# filter(DocType == "Journal") %>%
# group_by(PaperID) %>%
# filter(latam_prop >= 0.5 |
# any(AuthorSequenceNumber == 1 & is_latam == 1)) %>%
# ungroup()
#
## 4. Filtrar por tiempo
#rm(b3_joined)
#gc()
#b3_fromlatam_1990_2000 <- b3_fromlatam %>% filter(Year < 2000)Preparación de Datos
Siguiendo la conceptualización de Gondal sobre campos científicos emergentes, es crucial identificar y operacionalizar tres tipos de variables:
- Indicadores de centralización temprana (ej. productividad, citaciones)
- Medidas de diferenciación disciplinar (ej. campos STEM vs SHAPE)
- Métricas de disrupción que capturen la innovación en el campo
La siguiente preparación de datos organiza estas dimensiones:
Code
# Team_Size: Número de autores por paper
team_size_df <- b3_fromlatam_1990_2000 %>%
group_by(PaperID) %>%
summarise(
Team_Size = n_distinct(AuthorID)
)
# Combinar con el dataset original
b3_fromlatam_1990_2000 <- b3_fromlatam_1990_2000 %>%
left_join(team_size_df, by = "PaperID")
# 5. Preparación y limpieza
clean_data <- b3_fromlatam_1990_2000 %>%
filter(!is.na(Disruption),
!is.na(CitationCount),
!is.na(H.index_auth),
!is.na(Average_C10_auth),
!is.na(Productivity_auth),
!is.na(Affiliation_Name),
!is.na(is_latam),
!is.na(Institution_Count),
!is.na(Team_Size),
!is.na(Field_Name),
!is.na(Field_Type),
!is.na(name)) %>%
filter(Field_Type == "Top")
# 6. Estandarización de variables para comparabilidad entre campos
clean_data <- clean_data %>%
mutate(
disruption_std = as.vector(scale(Disruption)),
citations_std = as.vector(scale(log1p(CitationCount))),
h_index_std = as.vector(scale(log1p(H.index_auth))),
avg_c10_std = as.vector(scale(log1p(Average_C10_auth))),
productivity_std = as.vector(scale(log1p(Productivity_auth))),
# Clasificación disciplinar siguiendo la distinción
# teórica entre campos más y menos establecidos
field_broad = case_when(
Field_Name %in% c(
"Biology", "Chemistry", "Computer science",
"Engineering", "Environmental science", "Geography",
"Materials science", "Mathematics", "Medicine"
) ~ "STEM",
Field_Name %in% c(
"Business", "Economics", "Political science", "Sociology"
) ~ "SHAPE",
TRUE ~ NA_character_
)
)Construcción de la Red Bipartita
La perspectiva de Gondal sobre la dualidad estructural sugiere que la estructura del conocimiento emerge de la interacción entre dos modos: autores y documentos. Para capturar esta dualidad, construimos una red bipartita donde:
- El primer modo (papers) representa los vehículos de conocimiento
- El segundo modo (autores) representa los productores de conocimiento
- Los vínculos entre modos capturan las relaciones de autoría
Esta estructura nos permite examinar cómo las propiedades de cada modo contribuyen a la emergencia de patrones macro:
Code
# 7. Atributos por modo siguiendo la dualidad estructural
paper_attributes <- clean_data %>%
group_by(PaperID) %>%
slice(1) %>%
ungroup() %>%
select(PaperID, disruption_std, citations_std,
Team_Size, Institution_Count, Field_Name, field_broad)
author_attributes <- clean_data %>%
group_by(AuthorID) %>%
slice(1) %>%
ungroup()
# 8. Crear matriz de incidencia y red bipartita
papers <- unique(paper_attributes$PaperID)
authors <- unique(author_attributes$AuthorID)
# Matriz de incidencia que captura la dualidad autor-paper
paper_author_matrix <- sparseMatrix(
i = match(clean_data$PaperID, papers),
j = match(clean_data$AuthorID, authors),
x = 1,
dims = c(length(papers), length(authors))
)Construcción y Visualización de la Red
La visualización de redes bipartitas presenta desafíos particulares que Gondal (2011) aborda en su análisis. Es crucial representar visualmente las tres propiedades macro que teóricamente caracterizan los campos emergentes: estructura centro-periferia, cohesión estructural y características de mundo pequeño. Para esto, primero necesitamos establecer atributos que nos permitan identificar roles estructurales:
# Crear la red bipartita con los atributos necesarios
net_bipartite <- network(
paper_author_matrix,
matrix.type = "bipartite",
directed = FALSE
)
# Asignación de atributos siguiendo la teoría de Gondal
# Modo 1 (Papers): Vehículos de conocimiento
net_bipartite %v% "disruption" <- paper_attributes$disruption_std
net_bipartite %v% "citations" <- paper_attributes$citations_std
net_bipartite %v% "inst_count" <- paper_attributes$Institution_Count
net_bipartite %v% "team_size" <- paper_attributes$Team_Size
net_bipartite %v% "small_team" <- ifelse(net_bipartite %v% "team_size" <= 3, 1, 0)
net_bipartite %v% "field" <- paper_attributes$Field_Name
net_bipartite %v% "field_broad" <- paper_attributes$field_broad
# Modo 2 (Autores): Productores de conocimiento
net_bipartite %v% "h_index" <- author_attributes$h_index_std
net_bipartite %v% "avg_c10" <- author_attributes$avg_c10_std
net_bipartite %v% "affiliation" <- author_attributes$Affiliation_Name
net_bipartite %v% "is_latam" <- author_attributes$is_latam
net_bipartite %v% "country" <- author_attributes$name
net_bipartite %v% "productivity" <- author_attributes$productivity_stdVisualización de la Estructura Centro-Periferia
Siguiendo a Gondal, la estructura centro-periferia es especialmente relevante en campos emergentes. Para visualizarla, necesitamos primero identificar los roles estructurales de los nodos:
# Configuración de roles estructurales
n_vertices <- network.size(net_bipartite)
bipartite_value <- 161 # Número de papers
# Vector para identificar tipos de nodos
is_actor <- rep(FALSE, n_vertices)
is_actor[(bipartite_value + 1):n_vertices] <- TRUE
net_bipartite %v% "is_actor" <- is_actor
# Etiquetas para visualización
node_labels <- rep("Autor", n_vertices)
node_labels[1:bipartite_value] <- "Paper"
net_bipartite %v% "tipo" <- node_labels
# Calculamos grados para el objeto network
node_degrees <- degree(net_bipartite, gmode="graph")
scaled_degrees <- scales::rescale(node_degrees, to = c(2, 15))
# Asignamos los grados como atributo de vértice
net_bipartite %v% "node_size" <- scaled_degrees
pn <- ggraph(net_bipartite, layout = "graphopt") +
# Edges
geom_edge_link0(edge_colour = "black",
edge_width = 0.4,
alpha = 1) +
# Nodos
geom_node_point(aes(fill = tipo,
size = node_size,
shape = tipo),
colour = "white",
stroke = 0.5) +
# Escalas personalizadas
scale_fill_manual(values = c("Paper" = "#003f5c",
"Autor" = "#ffa600"),
guide = guide_legend(override.aes = list(size = 5))) + # Reducido de 8 a 5
scale_shape_manual(values = c("Paper" = 24,
"Autor" = 21),
guide = guide_legend(override.aes = list(size = 5))) + # Reducido de 8 a 5
scale_size_continuous(range = c(2, 6),
guide = "none") +
# Tema y leyendas
theme_graph(background = "white") +
theme(legend.position = "right",
legend.box.background = element_rect(color = "black", size = 0.3),
legend.key.size = unit(1, 'cm'), # Reducido de 1.5 a 1
legend.key.height = unit(0.8, 'cm'), # Reducido de 1.5 a 1
legend.key.width = unit(0.6, 'cm'), # Reducido de 1.5 a 1
legend.margin = margin(6, 6, 6, 6),
legend.text = element_text(size = 10),
plot.title = element_text(size = 14, face = "bold"),
plot.subtitle = element_text(size = 11)) +
# Etiquetas
labs(fill = "Tipo de Entidad",
shape = "Tipo de Entidad",
title = "",
subtitle = "")
#pn# Primero obtenemos las coordenadas usando layout stress
coords <- create_layout(net_bipartite, layout = "graphopt")
# Obtenemos la lista de bordes del objeto network de forma correcta
edges <- as.matrix.network.edgelist(net_bipartite)
# Creamos los datos para los nodos
nodes_df <- data.frame(
x = coords$x,
y = coords$y,
tipo = net_bipartite %v% "tipo",
size = net_bipartite %v% "node_size",
field = net_bipartite %v% "field_broad"
)
# Creamos los datos para los enlaces
edges_df <- data.frame(
x0 = coords$x[edges[,1]],
y0 = coords$y[edges[,1]],
x1 = coords$x[edges[,2]],
y1 = coords$y[edges[,2]]
)
# Crear el gráfico con plotly
p <- plot_ly() %>%
# Agregar los enlaces
add_segments(
data = edges_df,
x = ~x0,
y = ~y0,
xend = ~x1,
yend = ~y1,
line = list(color = "gray80", width = 0.5),
hoverinfo = "none",
showlegend = FALSE
) %>%
# Agregar los nodos
add_markers(
data = nodes_df,
x = ~x,
y = ~y,
size = ~size,
color = ~tipo,
colors = c("#003f5c", "#ffa600"),
symbol = ~tipo,
symbols = c("triangle-up", "circle"),
text = ~paste("Tipo:", tipo,
"<br>Field:", field,
"<br>Tamaño:", round(size, 2)),
hoverinfo = "text",
showlegend = TRUE
) %>%
# Configurar el layout
layout(
title = "",
showlegend = TRUE,
legend = list(orientation = "h", x = 0, y = -0.1),
xaxis = list(showgrid = FALSE, zeroline = FALSE, showticklabels = FALSE),
yaxis = list(showgrid = FALSE, zeroline = FALSE, showticklabels = FALSE),
hovermode = "closest",
dragmode = "pan"
)
pPropiedades Estructurales de la Red
Siguiendo a Gondal (2011), analizamos tres propiedades estructurales fundamentales de la red bipartita de colaboración científica:
Estadísticas Descriptivas Básicas
# Estadísticas básicas de la red
summary(net_bipartite, print.adj = FALSE)
# Densidad
network.density(net_bipartite)
# Distancia promedio y diámetro
geodist <- geodist(net_bipartite)
mean(geodist$gdist[geodist$gdist != Inf])
max(geodist$gdist[geodist$gdist != Inf])# Análisis de k-cores
kcores <- kcores(net_bipartite)
# Distribución de k-cores
table(kcores)kcores
2 4
422 21 # Componentes
components <- component.dist(net_bipartite)
table(components$csize)
2 3 4 5 6 7 9 15 22
67 34 22 7 4 2 1 1 1 # Crear tabla resumen de estadísticos de red
network_summary <- data.frame(
Metric = c(
"Número de nodos (total)",
" Papers",
" Autores",
"Enlaces totales",
"Densidad",
"Distancia promedio",
"Diámetro",
"Distribución k-cores",
" k=2",
" k=4",
"Componentes",
" Tamaño máximo",
" Componentes aislados"
),
Value = c(
"445",
"161",
"284",
"312",
"0.003",
"1.86",
"8",
"",
"424",
"21",
"",
"22",
"139"
),
Description = c(
"Total de vértices en la red",
"Artículos científicos",
"Investigadores",
"Vínculos de coautoría",
"Proporción de vínculos posibles realizados",
"Número promedio de pasos entre nodos",
"Máxima distancia entre dos nodos",
"Distribución de núcleos-k",
"Nodos con al menos 2 conexiones",
"Nodos con al menos 4 conexiones",
"Subgrupos conectados",
"Tamaño del componente más grande",
"Número de componentes desconectados"
)
)
# Mostrar tabla con formato
network_summary %>%
kable(col.names = c("Métrica", "Valor", "Descripción"),
caption = "Estadísticas Descriptivas de la Red") %>%
kable_classic_2(full_width = F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive")) %>%
pack_rows("Características Básicas", 1, 4) %>%
pack_rows("Medidas de Cohesión", 5, 7) %>%
pack_rows("Estructura Núcleo-Periferia", 8, 10) %>%
pack_rows("Fragmentación", 11, 13)| Métrica | Valor | Descripción |
|---|---|---|
| Características Básicas | ||
| Número de nodos (total) | 445 | Total de vértices en la red |
| Papers | 161 | Artículos científicos |
| Autores | 284 | Investigadores |
| Enlaces totales | 312 | Vínculos de coautoría |
| Medidas de Cohesión | ||
| Densidad | 0.003 | Proporción de vínculos posibles realizados |
| Distancia promedio | 1.86 | Número promedio de pasos entre nodos |
| Diámetro | 8 | Máxima distancia entre dos nodos |
| Estructura Núcleo-Periferia | ||
| Distribución k-cores | Distribución de núcleos-k | |
| k=2 | 424 | Nodos con al menos 2 conexiones |
| k=4 | 21 | Nodos con al menos 4 conexiones |
| Fragmentación | ||
| Componentes | Subgrupos conectados | |
| Tamaño máximo | 22 | Tamaño del componente más grande |
| Componentes aislados | 139 | Número de componentes desconectados |
Análisis de Distribución de Grados
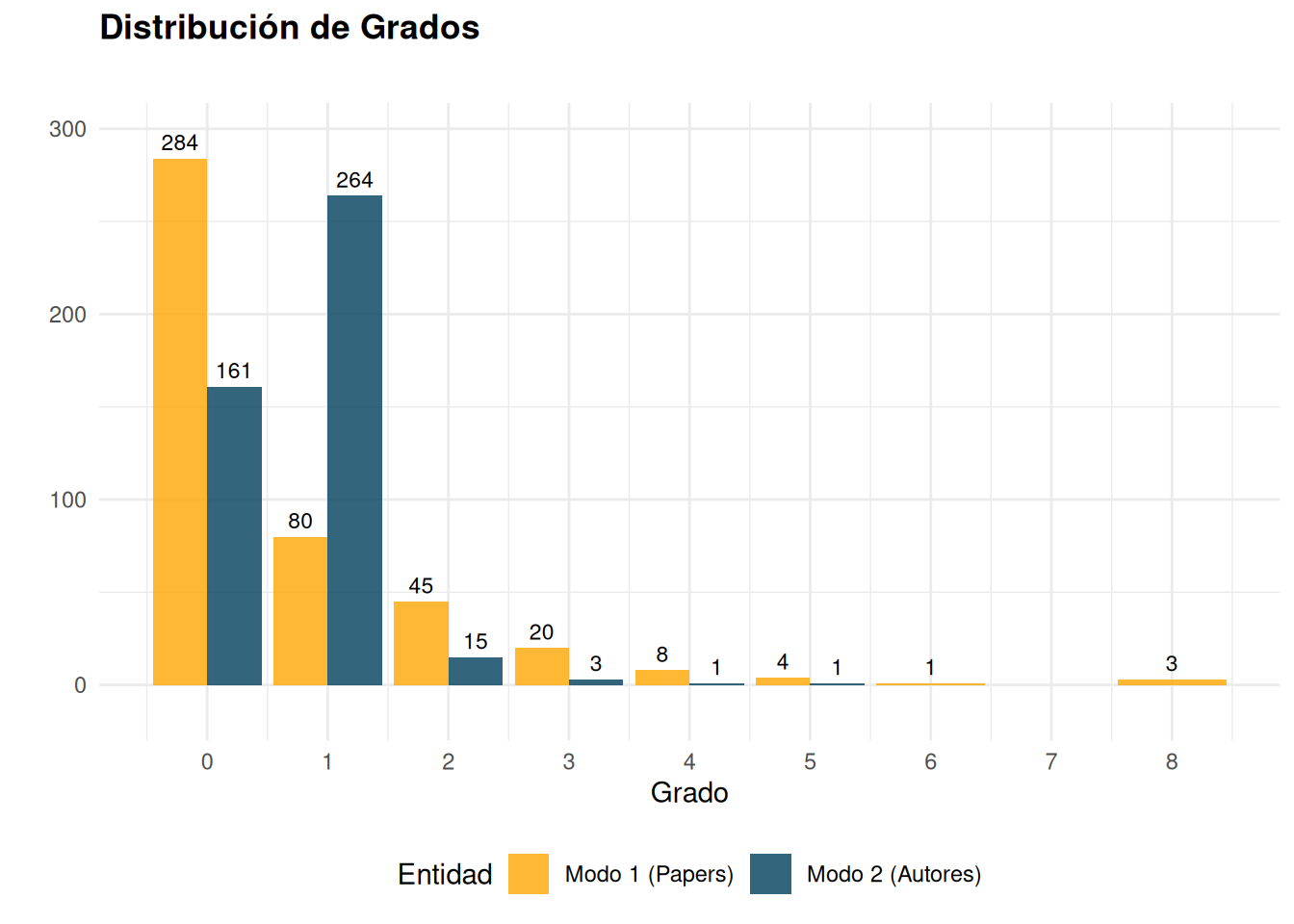
Gondal sugiere que en campos emergentes, la distribución de grados debería mostrar señales de centralización temprana. Específicamente, deberíamos observar:
- Una alta proporción de autores con pocos vínculos
- Un pequeño grupo de autores altamente conectados
- Patrones diferentes entre papers y autores
degreedist(net_bipartite)Bipartite mode 2 degree distribution:
0 1 2 3 4 5
161 262 15 3 1 1
Bipartite mode 1 degree distribution:
0 1 2 3 4 5 6 8
282 80 45 21 8 3 1 3 # Crear dataframes para cada modo
mode1_data <- data.frame(
grado = c(0, 1, 2, 3, 4, 5, 6, 8),
frecuencia = c(284, 80, 45, 20, 8, 4, 1, 3),
modo = "Modo 1 (Papers)"
)
mode2_data <- data.frame(
grado = c(0, 1, 2, 3, 4, 5),
frecuencia = c(161, 264, 15, 3, 1, 1),
modo = "Modo 2 (Autores)"
)
# Combinar los datos
degree_data <- rbind(mode1_data, mode2_data)
# Crear el gráfico
ggplot(degree_data, aes(x = grado, y = frecuencia, fill = modo)) +
geom_bar(stat = "identity", position = "dodge", alpha = 0.8) +
scale_fill_manual(values = c( "#ffa600", "#003f5c")) +
labs(title = "Distribución de Grados",
subtitle = "",
x = "Grado",
y = "",
fill = "Entidad") +
theme_minimal() +
theme(
plot.title = element_text(face = "bold"),
legend.position = "bottom"
) +
scale_x_continuous(breaks = 0:8) +
scale_y_continuous(expand = c(0, 30)) +
geom_text(aes(label = frecuencia),
position = position_dodge(width = 0.9),
vjust = -0.5,
size = 3)# Para las métricas, usamos funciones de sna
bipartite_metrics <- data.frame(
node = network.vertex.names(net_bipartite),
tipo = net_bipartite %v% "tipo",
degree = degree(net_bipartite, gmode="graph"),
betweenness = betweenness(net_bipartite),
eigenvector = evcent(net_bipartite)
) %>%
group_by(tipo) %>%
summarise(
avg_degree = mean(degree),
max_degree = max(degree),
avg_betweenness = mean(betweenness),
avg_eigenvector = mean(eigenvector)
)Warning in evcent(net_bipartite): Maximum iterations exceeded in evcent_R
without convergence. This matrix may be pathological - increase maxiter or try
eigen().kable(as_tibble(bipartite_metrics), caption = "Resumen centralidad por entidad") %>%
kable_classic_2(full_width = F) %>%
kable_styling(bootstrap_options = c("striped", "hover", "condensed", "responsive"))| tipo | avg_degree | max_degree | avg_betweenness | avg_eigenvector |
|---|---|---|---|---|
| Autor | 1.099291 | 5 | 2.829787 | 0.0087002 |
| Paper | 1.925466 | 8 | 9.279503 | 0.0060304 |
Análisis ERGM: Modelando Mecanismos Generativos
Siguiendo a Gondal (2011), el modelamiento ERGM nos permite examinar los mecanismos micro que generan las estructuras macro observadas. Desarrollamos una estrategia de modelamiento en cuatro etapas, cada una capturando diferentes aspectos teóricos:
- Modelo Base: Examina la tendencia base a formar vínculos y el efecto de los campos disciplinares
- Modelo de Grados: Incorpora la geometría ponderada de grados para capturar centralización
- Modelo con Covariables: Agrega atributos nodales como disrupción y productividad
- Modelo Final: Integra efectos de interacción y homofilia disciplinar
# Usar el 75% de los núcleos disponibles
num_cores <- parallel::detectCores() - 1
# Si quieres ver cuántos núcleos se usarán
cat("Usando", num_cores, "de", parallel::detectCores(), "núcleos disponibles\n")Usando 15 de 16 núcleos disponibles# 1. Modelo simple (mantenemos igual como base)
model_simple <- ergm(
net_bipartite ~ edges + b1factor("field_broad"),
control = control.ergm(
MCMLE.maxit = 5,
MCMC.samplesize = 1000,
MCMLE.termination = "Hummel"
)
)
# 2. Modelo con términos de grado (mantenemos igual)
model_degrees <- ergm(
net_bipartite ~
edges +
gwb1degree(decay = 0.25, fixed = T) +
gwb2degree(decay = 0.25, fixed = T) +
b1factor("field_broad"),
control = control.ergm(
init = c(coef(model_simple), rep(0, 2)),
MCMLE.maxit = 15,
MCMC.samplesize = 2000,
MCMLE.termination = "Hummel"
)
)Warning: 'glpk' selected as the solver, but package 'Rglpk' is not available;
falling back to 'lpSolveAPI'. This should be fine unless the sample size and/or
the number of parameters is very big.# 3. Modelo con covariables (mantenemos igual)
model_covars <- ergm(
net_bipartite ~
edges +
gwb1degree(decay = 0.25, fixed = T) +
gwb2degree(decay = 0.25, fixed = T) +
b1factor("field_broad") +
b1cov("disruption") +
b2cov("productivity"),
control = control.ergm(
init = c(coef(model_degrees), rep(0, 2)),
MCMLE.maxit = 20,
MCMC.samplesize = 3000,
MCMLE.termination = "Hummel"
)
)
num_params_simple <- length(coef(model_simple))
num_params_degrees <- length(coef(model_degrees))
num_params_covars <- length(coef(model_covars))
# Calculamos el número de parámetros adicionales para el modelo final
num_fields <- length(unique(net_bipartite %v% "field_broad"))
additional_params <- num_fields + 1 # Para nodematch con diff=TRUE y country
# Ajustamos el modelo final
model_final <- ergm(
net_bipartite ~
edges +
gwb1degree(decay = 0.25, fixed = T) +
gwb2degree(decay = 0.25, fixed = T) +
b1cov("disruption") +
b2cov("productivity") +
nodematch("field_broad", diff = TRUE) +
nodematch("country") +
b1cov("disruption"):b2cov("productivity"),
control = control.ergm(
init = c(coef(model_covars), rep(0, additional_params)),
MCMLE.maxit = 30,
MCMC.burnin = 75000,
MCMC.interval = 1500,
MCMC.samplesize = 12500,
MCMLE.density.guard = 40,
MCMLE.sequential = TRUE,
MCMLE.steplength = 1,
MCMLE.steplength.margin = 0.5,
MCMLE.termination = "Hummel",
parallel = num_cores,
parallel.type = "PSOCK",
seed = 123
)
)summary(model_final)Call:
ergm(formula = net_bipartite ~ edges + gwb1degree(decay = 0.25,
fixed = T) + gwb2degree(decay = 0.25, fixed = T) + b1cov("disruption") +
b2cov("productivity") + nodematch("field_broad", diff = TRUE) +
nodematch("country") + b1cov("disruption"):b2cov("productivity"),
control = control.ergm(init = c(coef(model_covars), rep(0,
additional_params)), MCMLE.maxit = 30, MCMC.burnin = 75000,
MCMC.interval = 1500, MCMC.samplesize = 12500, MCMLE.density.guard = 40,
MCMLE.sequential = TRUE, MCMLE.steplength = 1, MCMLE.steplength.margin = 0.5,
MCMLE.termination = "Hummel", parallel = num_cores, parallel.type = "PSOCK",
seed = 123))
Monte Carlo Maximum Likelihood Results:
Estimate Std. Error MCMC % z value Pr(>|z|)
edges -9.13164 0.36625 0 -24.933 <1e-04
gwb1deg.fixed.0.25 3.01132 0.58220 0 5.172 <1e-04
gwb2deg.fixed.0.25 8.50913 0.97582 0 8.720 <1e-04
b1cov.disruption 0.02342 0.07170 0 0.327 0.744
b2cov.productivity -0.09787 0.19032 0 -0.514 0.607
nodematch.field_broad.SHAPE -0.81809 0.20671 0 -3.958 <1e-04
nodematch.field_broad.STEM 0.89647 0.18610 0 4.817 <1e-04
nodematch.country -0.06518 0.15239 1 -0.428 0.669
b1cov.disruption:b2cov.productivity -0.01569 0.04901 1 -0.320 0.749
edges ***
gwb1deg.fixed.0.25 ***
gwb2deg.fixed.0.25 ***
b1cov.disruption
b2cov.productivity
nodematch.field_broad.SHAPE ***
nodematch.field_broad.STEM ***
nodematch.country
b1cov.disruption:b2cov.productivity
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Null Deviance: 62941 on 45402 degrees of freedom
Residual Deviance: 3263 on 45393 degrees of freedom
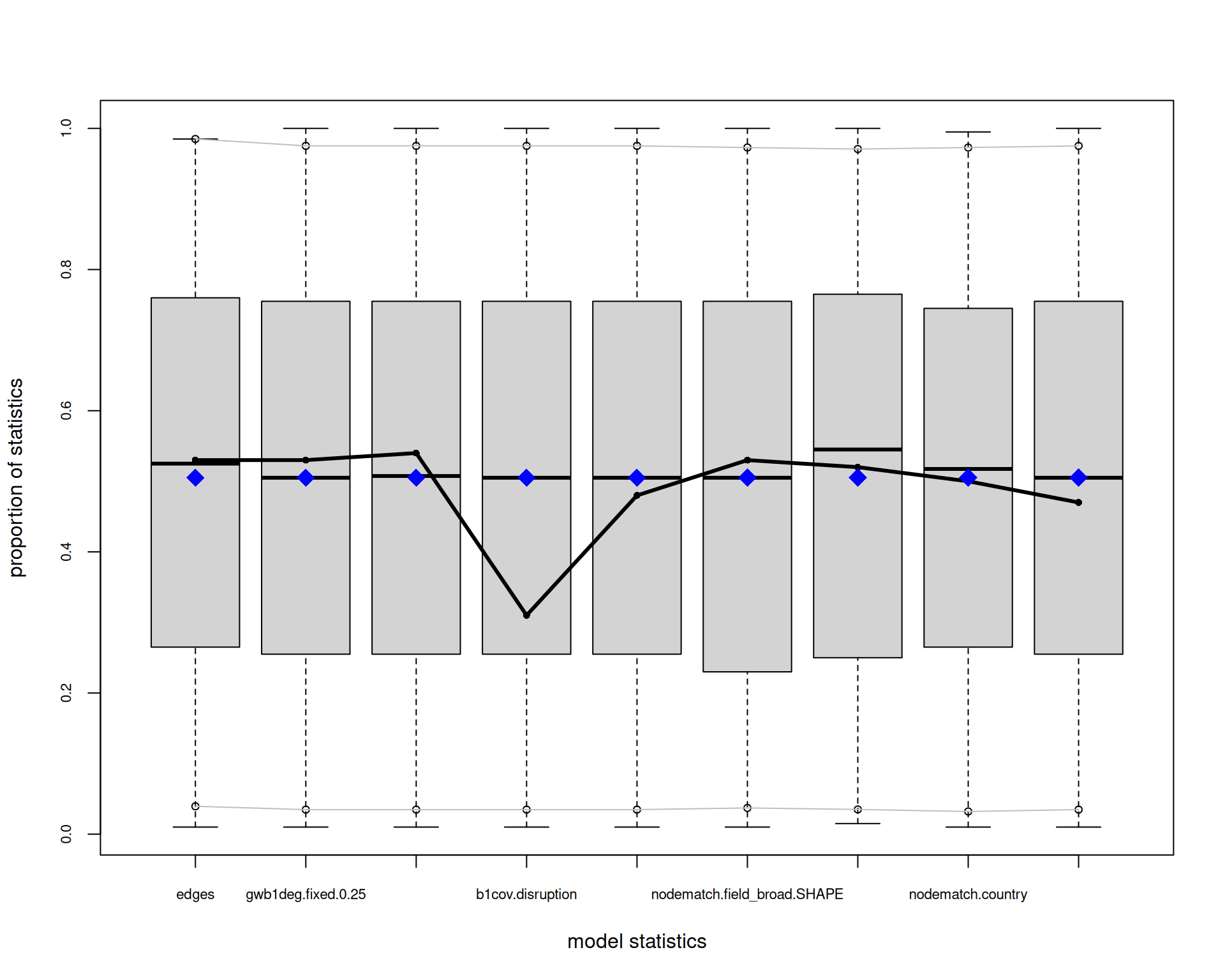
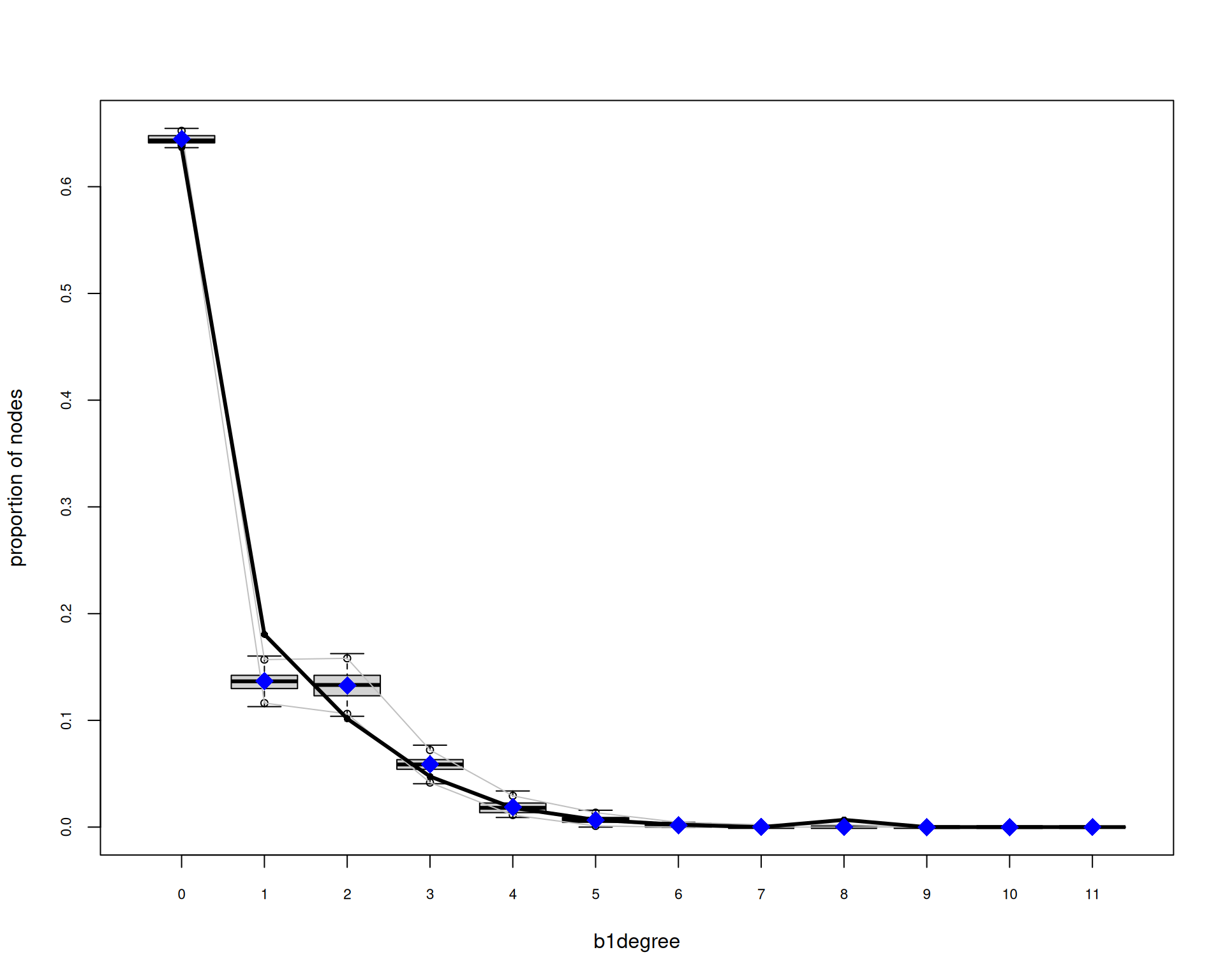
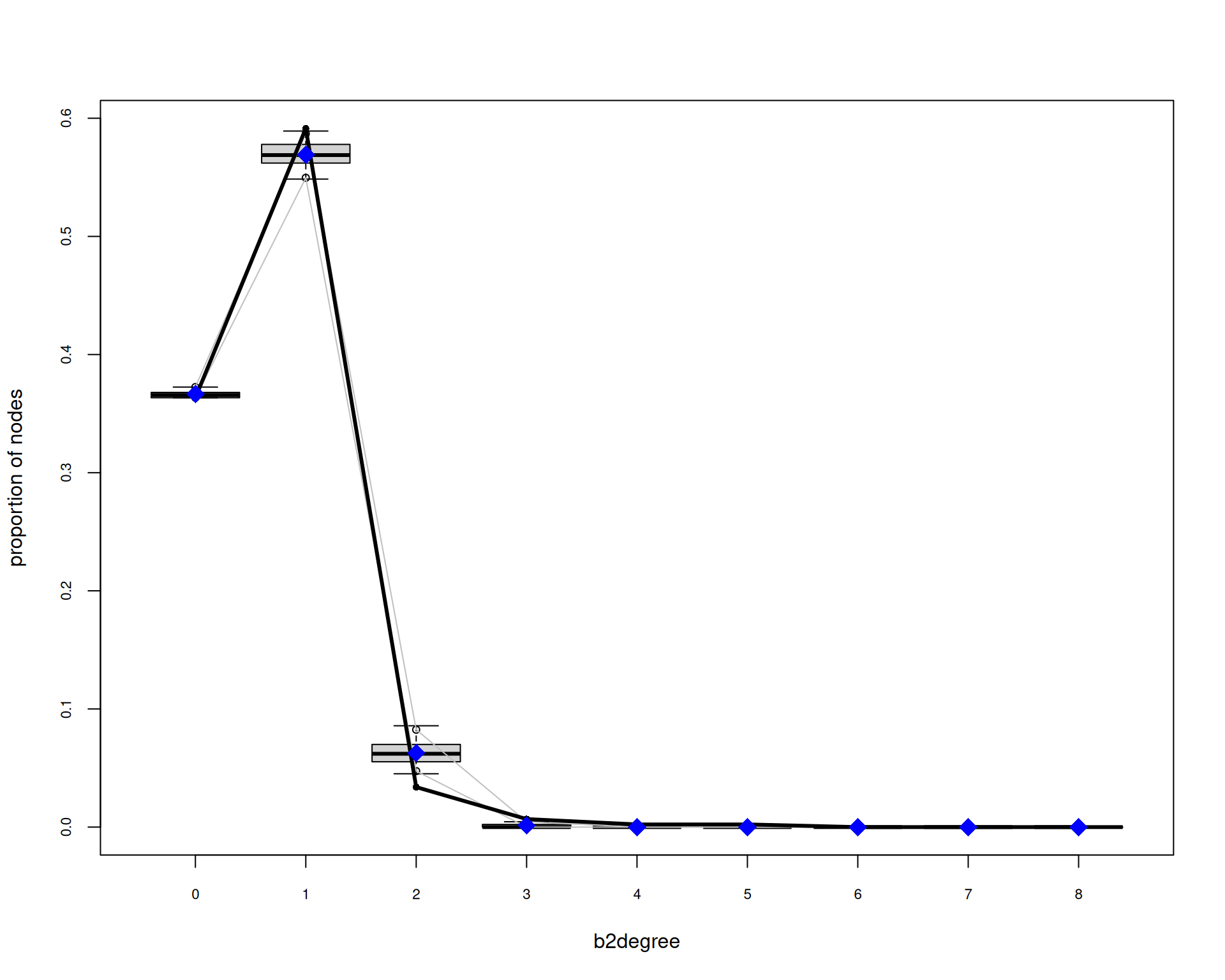
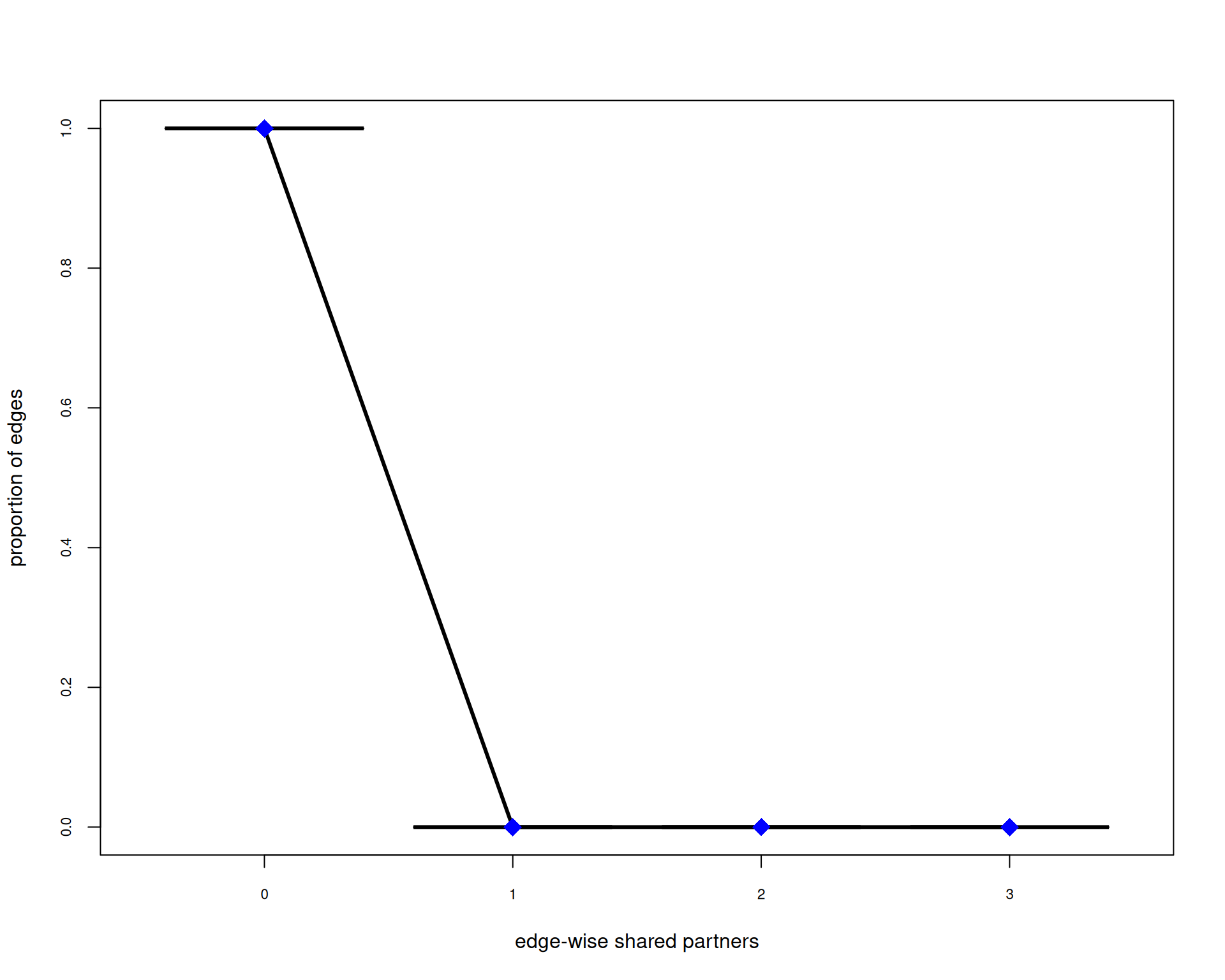
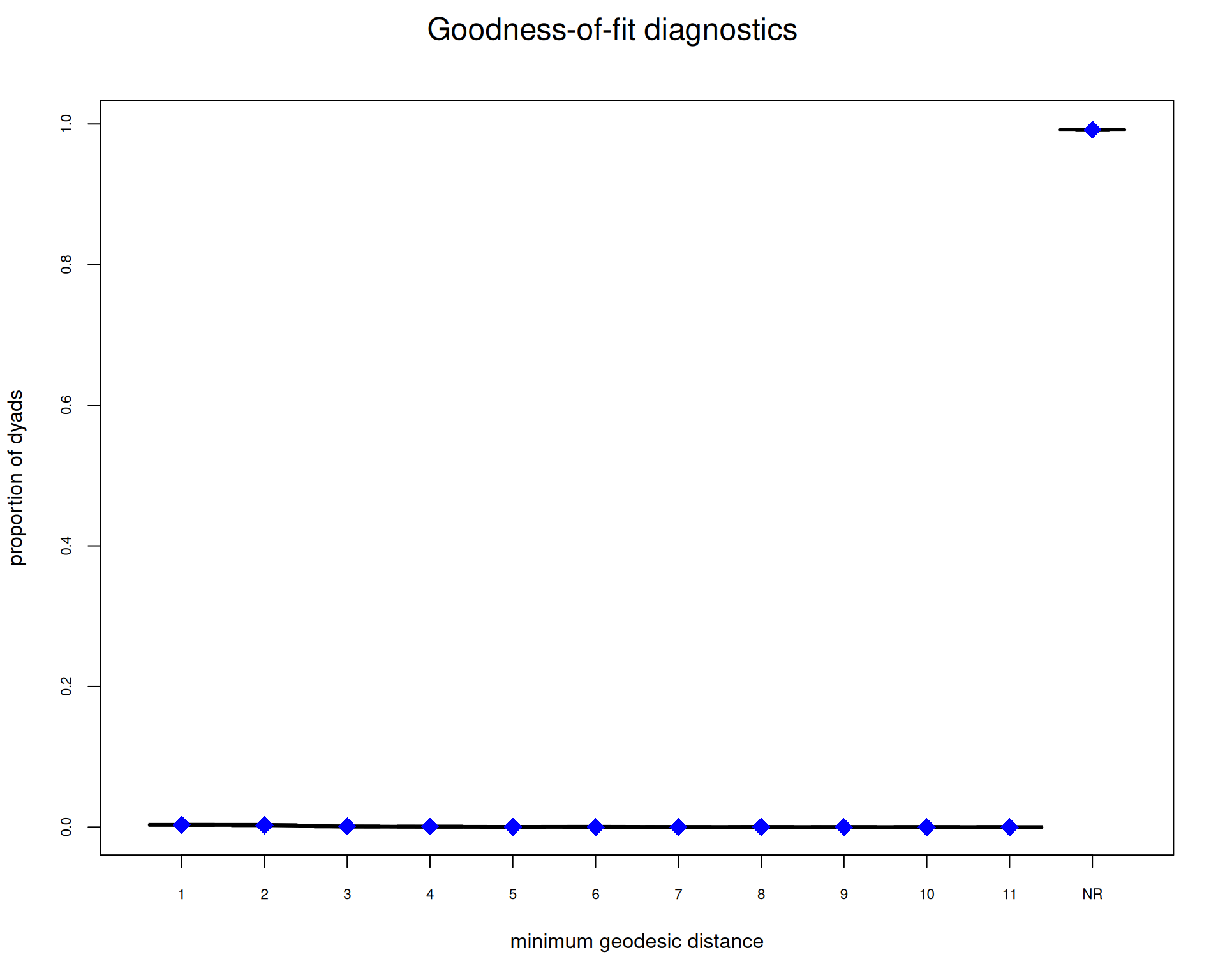
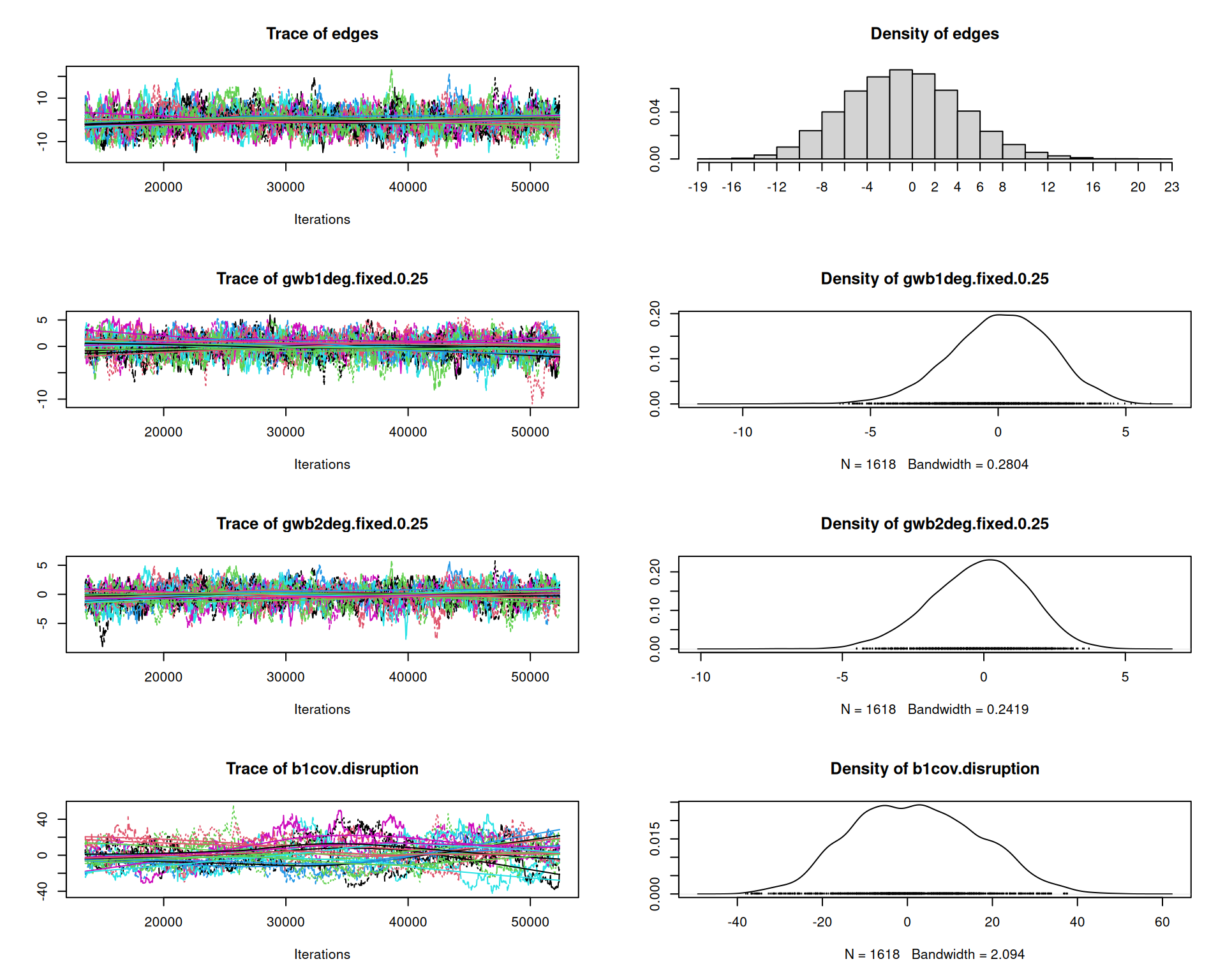
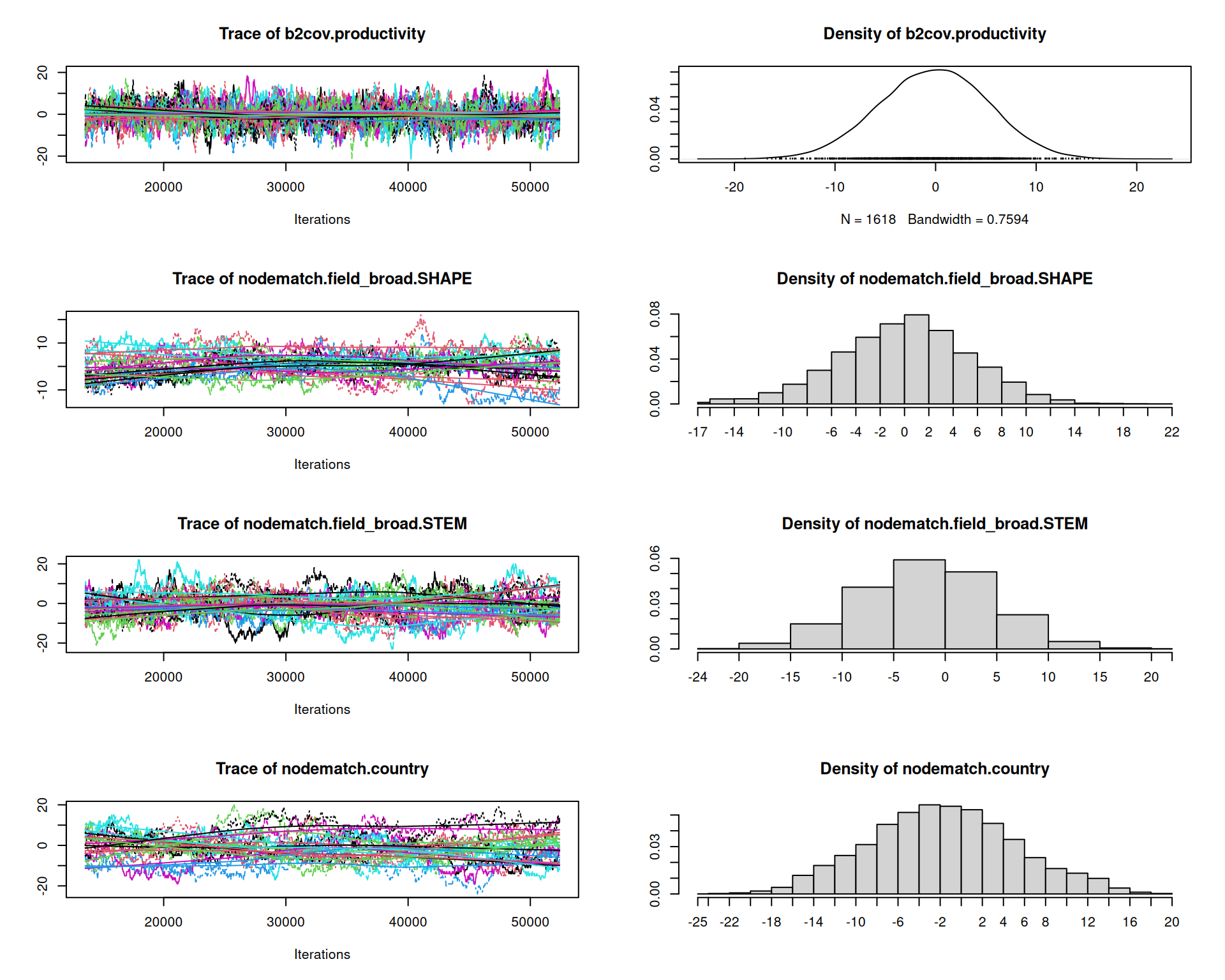
AIC: 3281 BIC: 3360 (Smaller is better. MC Std. Err. = 2.472)Diagnósticos del Modelo
# MCMC diagnósticos
mcmc.diagnostics(model_final)Sample statistics summary:
Iterations = 13584:52392
Thinning interval = 24
Number of chains = 15
Sample size per chain = 1618
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
edges -0.2820 5.095 0.03271 0.15926
gwb1deg.fixed.0.25 0.1083 1.993 0.01279 0.07156
gwb2deg.fixed.0.25 -0.1573 1.720 0.01104 0.06292
b1cov.disruption 1.9908 14.883 0.09553 1.15998
b2cov.productivity -0.1768 5.398 0.03465 0.17429
nodematch.field_broad.SHAPE 0.4475 5.422 0.03480 0.31960
nodematch.field_broad.STEM -1.2785 6.350 0.04076 0.36813
nodematch.country -1.2175 6.962 0.04469 0.49259
b1cov.disruption:b2cov.productivity -5.0231 21.311 0.13679 2.34273
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
edges -10.000 -4.000 0.00000 3.000 10.000
gwb1deg.fixed.0.25 -4.004 -1.175 0.18412 1.504 3.793
gwb2deg.fixed.0.25 -3.729 -1.278 -0.05669 1.049 2.934
b1cov.disruption -24.850 -9.042 1.52899 12.660 30.881
b2cov.productivity -10.843 -3.803 -0.11106 3.532 10.257
nodematch.field_broad.SHAPE -11.000 -3.000 1.00000 4.000 11.000
nodematch.field_broad.STEM -14.000 -6.000 -1.00000 3.000 11.000
nodematch.country -14.000 -6.000 -1.00000 3.000 13.000
b1cov.disruption:b2cov.productivity -47.109 -19.659 -5.22234 9.608 37.536
Sample statistics cross-correlations:
edges gwb1deg.fixed.0.25
edges 1.00000000 0.35717947
gwb1deg.fixed.0.25 0.35717947 1.00000000
gwb2deg.fixed.0.25 0.79631871 0.27424263
b1cov.disruption -0.06451633 -0.01287898
b2cov.productivity -0.21956849 -0.06012029
nodematch.field_broad.SHAPE 0.08288193 0.23135286
nodematch.field_broad.STEM 0.33262472 -0.09282215
nodematch.country 0.08112202 0.01171890
b1cov.disruption:b2cov.productivity 0.03929605 -0.03308404
gwb2deg.fixed.0.25 b1cov.disruption
edges 0.79631871 -0.064516332
gwb1deg.fixed.0.25 0.27424263 -0.012878976
gwb2deg.fixed.0.25 1.00000000 -0.031169331
b1cov.disruption -0.03116933 1.000000000
b2cov.productivity -0.16150782 0.008359676
nodematch.field_broad.SHAPE 0.06273395 -0.103380241
nodematch.field_broad.STEM 0.18356358 -0.042671804
nodematch.country 0.06259831 0.198862982
b1cov.disruption:b2cov.productivity 0.02956973 -0.200075287
b2cov.productivity
edges -0.219568489
gwb1deg.fixed.0.25 -0.060120294
gwb2deg.fixed.0.25 -0.161507817
b1cov.disruption 0.008359676
b2cov.productivity 1.000000000
nodematch.field_broad.SHAPE -0.033851165
nodematch.field_broad.STEM -0.106856529
nodematch.country -0.002789855
b1cov.disruption:b2cov.productivity 0.019149117
nodematch.field_broad.SHAPE
edges 0.08288193
gwb1deg.fixed.0.25 0.23135286
gwb2deg.fixed.0.25 0.06273395
b1cov.disruption -0.10338024
b2cov.productivity -0.03385116
nodematch.field_broad.SHAPE 1.00000000
nodematch.field_broad.STEM 0.27037517
nodematch.country 0.14964864
b1cov.disruption:b2cov.productivity -0.10512505
nodematch.field_broad.STEM
edges 0.332624721
gwb1deg.fixed.0.25 -0.092822154
gwb2deg.fixed.0.25 0.183563582
b1cov.disruption -0.042671804
b2cov.productivity -0.106856529
nodematch.field_broad.SHAPE 0.270375175
nodematch.field_broad.STEM 1.000000000
nodematch.country 0.166305081
b1cov.disruption:b2cov.productivity 0.007956778
nodematch.country
edges 0.081122016
gwb1deg.fixed.0.25 0.011718896
gwb2deg.fixed.0.25 0.062598314
b1cov.disruption 0.198862982
b2cov.productivity -0.002789855
nodematch.field_broad.SHAPE 0.149648638
nodematch.field_broad.STEM 0.166305081
nodematch.country 1.000000000
b1cov.disruption:b2cov.productivity 0.047352721
b1cov.disruption:b2cov.productivity
edges 0.039296050
gwb1deg.fixed.0.25 -0.033084040
gwb2deg.fixed.0.25 0.029569734
b1cov.disruption -0.200075287
b2cov.productivity 0.019149117
nodematch.field_broad.SHAPE -0.105125053
nodematch.field_broad.STEM 0.007956778
nodematch.country 0.047352721
b1cov.disruption:b2cov.productivity 1.000000000
Sample statistics auto-correlation:
Chain 1
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9120925 0.9233802 0.9341439 0.9859549
Lag 48 0.8310270 0.8514805 0.8729996 0.9719618
Lag 72 0.7597058 0.7902043 0.8165169 0.9590313
Lag 96 0.6945681 0.7367742 0.7644676 0.9467862
Lag 120 0.6333380 0.6863328 0.7151088 0.9351396
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9205295 0.9759446
Lag 48 0.8469365 0.9524856
Lag 72 0.7852171 0.9302886
Lag 96 0.7252309 0.9107950
Lag 120 0.6756432 0.8928120
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9827062 0.9895188
Lag 48 0.9668271 0.9791120
Lag 72 0.9526215 0.9690422
Lag 96 0.9383799 0.9592943
Lag 120 0.9254771 0.9499381
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9819164
Lag 48 0.9634959
Lag 72 0.9447102
Lag 96 0.9268402
Lag 120 0.9084457
Chain 2
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9032363 0.9082682 0.9356672 0.9802950
Lag 48 0.8209613 0.8311427 0.8794938 0.9629846
Lag 72 0.7455600 0.7696330 0.8237378 0.9456750
Lag 96 0.6752781 0.7195763 0.7729157 0.9297670
Lag 120 0.6158258 0.6780195 0.7268165 0.9157132
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9235116 0.9899214
Lag 48 0.8518740 0.9799658
Lag 72 0.7863462 0.9699580
Lag 96 0.7323868 0.9610792
Lag 120 0.6873819 0.9523854
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9670802 0.9847338
Lag 48 0.9380701 0.9694165
Lag 72 0.9099712 0.9546821
Lag 96 0.8833521 0.9403062
Lag 120 0.8606896 0.9262611
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9816737
Lag 48 0.9663535
Lag 72 0.9504536
Lag 96 0.9359600
Lag 120 0.9219376
Chain 3
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.8997270 0.9150878 0.9385347 0.9877465
Lag 48 0.8082549 0.8376270 0.8838730 0.9754437
Lag 72 0.7288088 0.7694756 0.8347343 0.9625881
Lag 96 0.6594711 0.7064035 0.7943434 0.9509525
Lag 120 0.5965423 0.6498121 0.7545578 0.9404866
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9128859 0.9720337
Lag 48 0.8300345 0.9458632
Lag 72 0.7504890 0.9193530
Lag 96 0.6735303 0.8933766
Lag 120 0.6031456 0.8667207
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9600022 0.9763950
Lag 48 0.9258025 0.9536531
Lag 72 0.8968491 0.9321460
Lag 96 0.8697552 0.9131724
Lag 120 0.8439370 0.8952798
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9925654
Lag 48 0.9851475
Lag 72 0.9778884
Lag 96 0.9708565
Lag 120 0.9634778
Chain 4
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9166772 0.9201679 0.9327681 0.9810231
Lag 48 0.8338302 0.8510864 0.8673720 0.9624592
Lag 72 0.7597512 0.7903353 0.8114988 0.9458075
Lag 96 0.7009140 0.7359605 0.7659072 0.9309080
Lag 120 0.6412987 0.6895100 0.7169862 0.9158271
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9294540 0.9763627
Lag 48 0.8601703 0.9516183
Lag 72 0.7950301 0.9270380
Lag 96 0.7283416 0.9028383
Lag 120 0.6644680 0.8812718
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9607256 0.9853004
Lag 48 0.9231227 0.9710192
Lag 72 0.8885032 0.9574632
Lag 96 0.8580840 0.9444623
Lag 120 0.8277803 0.9302870
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9963304
Lag 48 0.9926391
Lag 72 0.9889926
Lag 96 0.9855934
Lag 120 0.9823139
Chain 5
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9024652 0.9174738 0.9294966 0.9905350
Lag 48 0.8142157 0.8404586 0.8673511 0.9808088
Lag 72 0.7337196 0.7751333 0.8083485 0.9713747
Lag 96 0.6595755 0.7108923 0.7503666 0.9624511
Lag 120 0.5910376 0.6587873 0.6938159 0.9535428
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9233527 0.9757506
Lag 48 0.8576632 0.9524677
Lag 72 0.7974640 0.9333347
Lag 96 0.7344043 0.9141603
Lag 120 0.6801225 0.8983349
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9794083 0.9917159
Lag 48 0.9606140 0.9828039
Lag 72 0.9441768 0.9744647
Lag 96 0.9264838 0.9666495
Lag 120 0.9092373 0.9604134
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9897599
Lag 48 0.9795072
Lag 72 0.9699152
Lag 96 0.9604125
Lag 120 0.9505502
Chain 6
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9088073 0.9373028 0.9250854 0.9921552
Lag 48 0.8275058 0.8794522 0.8541048 0.9842967
Lag 72 0.7524681 0.8273034 0.7843437 0.9772435
Lag 96 0.6866523 0.7781508 0.7185894 0.9694015
Lag 120 0.6216059 0.7322106 0.6531833 0.9618611
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9339352 0.9763753
Lag 48 0.8684446 0.9548989
Lag 72 0.8083041 0.9336040
Lag 96 0.7500482 0.9126758
Lag 120 0.6920445 0.8924784
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9697076 0.9908360
Lag 48 0.9379438 0.9822048
Lag 72 0.9086710 0.9743052
Lag 96 0.8823636 0.9672169
Lag 120 0.8566748 0.9599773
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9856746
Lag 48 0.9712134
Lag 72 0.9582466
Lag 96 0.9459363
Lag 120 0.9363315
Chain 7
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9251345 0.9090246 0.9526552 0.9876914
Lag 48 0.8567616 0.8278131 0.9088931 0.9766238
Lag 72 0.7978654 0.7654600 0.8675798 0.9657021
Lag 96 0.7449280 0.7099760 0.8277603 0.9551226
Lag 120 0.6952003 0.6545899 0.7919698 0.9456996
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9243091 0.9758267
Lag 48 0.8558702 0.9528779
Lag 72 0.7943237 0.9321449
Lag 96 0.7349291 0.9103692
Lag 120 0.6736641 0.8897409
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9650816 0.9703449
Lag 48 0.9320306 0.9424234
Lag 72 0.9007774 0.9160692
Lag 96 0.8741842 0.8891192
Lag 120 0.8493101 0.8636531
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9880415
Lag 48 0.9752636
Lag 72 0.9622665
Lag 96 0.9497430
Lag 120 0.9385078
Chain 8
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9186868 0.9596794 0.9371897 0.9894946
Lag 48 0.8476896 0.9250755 0.8791173 0.9797641
Lag 72 0.7855985 0.8905003 0.8245959 0.9696468
Lag 96 0.7215353 0.8595119 0.7721712 0.9594202
Lag 120 0.6621198 0.8342866 0.7247936 0.9500163
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9323042 0.9845940
Lag 48 0.8716520 0.9694723
Lag 72 0.8122558 0.9547178
Lag 96 0.7550049 0.9414441
Lag 120 0.7024767 0.9283693
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9762976 0.9896974
Lag 48 0.9541189 0.9802743
Lag 72 0.9335740 0.9718455
Lag 96 0.9130670 0.9644110
Lag 120 0.8951106 0.9578313
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9946338
Lag 48 0.9893638
Lag 72 0.9842475
Lag 96 0.9791291
Lag 120 0.9737509
Chain 9
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9205294 0.9289431 0.9329035 0.9761885
Lag 48 0.8489632 0.8652908 0.8735210 0.9535336
Lag 72 0.7900180 0.8087985 0.8206842 0.9311834
Lag 96 0.7294912 0.7531167 0.7698254 0.9115103
Lag 120 0.6737382 0.7051920 0.7245494 0.8933189
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9200242 0.9700722
Lag 48 0.8424448 0.9427258
Lag 72 0.7744342 0.9168385
Lag 96 0.7113383 0.8899056
Lag 120 0.6507180 0.8652686
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9700210 0.9925940
Lag 48 0.9414730 0.9855069
Lag 72 0.9143517 0.9785225
Lag 96 0.8890629 0.9712901
Lag 120 0.8633595 0.9639867
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9910730
Lag 48 0.9826074
Lag 72 0.9746395
Lag 96 0.9669360
Lag 120 0.9594711
Chain 10
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9159563 0.9339961 0.9397713 0.9899860
Lag 48 0.8457124 0.8804003 0.8880121 0.9796389
Lag 72 0.7828039 0.8352158 0.8365694 0.9695737
Lag 96 0.7274755 0.7986351 0.7939152 0.9594294
Lag 120 0.6765955 0.7650567 0.7560744 0.9496866
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9329901 0.9914645
Lag 48 0.8720943 0.9836452
Lag 72 0.8160380 0.9758239
Lag 96 0.7624993 0.9689667
Lag 120 0.7141924 0.9626247
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9497787 0.9895138
Lag 48 0.9080289 0.9797835
Lag 72 0.8725089 0.9704753
Lag 96 0.8401962 0.9617793
Lag 120 0.8103672 0.9532643
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9962587
Lag 48 0.9925353
Lag 72 0.9887710
Lag 96 0.9849763
Lag 120 0.9810789
Chain 11
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9321339 0.9243001 0.9495301 0.9878334
Lag 48 0.8721184 0.8533698 0.9028221 0.9758325
Lag 72 0.8150525 0.7834645 0.8573530 0.9642395
Lag 96 0.7620616 0.7177108 0.8143068 0.9540668
Lag 120 0.7125496 0.6595805 0.7750807 0.9437661
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9419219 0.9771455
Lag 48 0.8850131 0.9553088
Lag 72 0.8272499 0.9340032
Lag 96 0.7730225 0.9156921
Lag 120 0.7263383 0.8967615
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9746538 0.9903193
Lag 48 0.9513743 0.9815430
Lag 72 0.9303454 0.9732020
Lag 96 0.9113544 0.9649542
Lag 120 0.8937577 0.9574709
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9913005
Lag 48 0.9820857
Lag 72 0.9726118
Lag 96 0.9638702
Lag 120 0.9557559
Chain 12
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9286804 0.9117724 0.9460670 0.9796224
Lag 48 0.8606319 0.8306505 0.8976407 0.9584215
Lag 72 0.8011885 0.7548441 0.8494690 0.9387358
Lag 96 0.7403356 0.6810190 0.8035819 0.9192334
Lag 120 0.6826200 0.6097556 0.7593916 0.9007375
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9223018 0.9775042
Lag 48 0.8485268 0.9557263
Lag 72 0.7770357 0.9330729
Lag 96 0.7071191 0.9127388
Lag 120 0.6356359 0.8939534
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9637303 0.9791499
Lag 48 0.9309020 0.9578666
Lag 72 0.9014865 0.9374525
Lag 96 0.8726883 0.9178045
Lag 120 0.8453295 0.8978884
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9926179
Lag 48 0.9853504
Lag 72 0.9781145
Lag 96 0.9705172
Lag 120 0.9629661
Chain 13
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9104635 0.9117029 0.9188207 0.9879366
Lag 48 0.8284935 0.8270433 0.8411267 0.9759987
Lag 72 0.7512067 0.7503428 0.7696121 0.9632879
Lag 96 0.6833904 0.6810921 0.7030756 0.9508535
Lag 120 0.6208959 0.6200330 0.6433673 0.9380461
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9218710 0.9815423
Lag 48 0.8472295 0.9626504
Lag 72 0.7804735 0.9449428
Lag 96 0.7253534 0.9276071
Lag 120 0.6662514 0.9114559
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9811748 0.9849153
Lag 48 0.9654473 0.9693845
Lag 72 0.9503962 0.9542999
Lag 96 0.9364865 0.9391367
Lag 120 0.9247124 0.9253011
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9887161
Lag 48 0.9781238
Lag 72 0.9678920
Lag 96 0.9582804
Lag 120 0.9485591
Chain 14
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9127116 0.9109397 0.9444760 0.9857821
Lag 48 0.8425846 0.8293000 0.8970878 0.9716230
Lag 72 0.7727509 0.7421247 0.8478138 0.9581832
Lag 96 0.7101217 0.6716141 0.8017791 0.9448542
Lag 120 0.6547228 0.6055890 0.7575544 0.9325802
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9225694 0.9780275
Lag 48 0.8532926 0.9562117
Lag 72 0.7876055 0.9345579
Lag 96 0.7287768 0.9156793
Lag 120 0.6742238 0.8979257
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9654760 0.9801869
Lag 48 0.9361689 0.9611194
Lag 72 0.9065661 0.9443978
Lag 96 0.8780659 0.9296038
Lag 120 0.8501824 0.9139732
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9915790
Lag 48 0.9836696
Lag 72 0.9762874
Lag 96 0.9696772
Lag 120 0.9637039
Chain 15
edges gwb1deg.fixed.0.25 gwb2deg.fixed.0.25 b1cov.disruption
Lag 0 1.0000000 1.0000000 1.0000000 1.0000000
Lag 24 0.9407548 0.9365542 0.9436062 0.9799980
Lag 48 0.8841027 0.8827926 0.8879250 0.9606316
Lag 72 0.8305117 0.8303017 0.8320161 0.9424400
Lag 96 0.7869362 0.7868485 0.7834451 0.9271901
Lag 120 0.7452119 0.7486166 0.7340387 0.9118221
b2cov.productivity nodematch.field_broad.SHAPE
Lag 0 1.0000000 1.0000000
Lag 24 0.9182157 0.9771615
Lag 48 0.8385892 0.9552291
Lag 72 0.7613916 0.9343386
Lag 96 0.6913044 0.9174557
Lag 120 0.6239253 0.9025631
nodematch.field_broad.STEM nodematch.country
Lag 0 1.0000000 1.0000000
Lag 24 0.9783502 0.9842600
Lag 48 0.9567289 0.9685680
Lag 72 0.9359584 0.9550082
Lag 96 0.9175764 0.9406553
Lag 120 0.8990156 0.9258288
b1cov.disruption:b2cov.productivity
Lag 0 1.0000000
Lag 24 0.9953659
Lag 48 0.9906049
Lag 72 0.9857772
Lag 96 0.9810534
Lag 120 0.9767152
Sample statistics burn-in diagnostic (Geweke):
Chain 1
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
-1.0889227 1.2177152
gwb2deg.fixed.0.25 b1cov.disruption
-0.6076943 -0.7176758
b2cov.productivity nodematch.field_broad.SHAPE
0.2009061 -1.9213887
nodematch.field_broad.STEM nodematch.country
1.5965246 6.3287404
b1cov.disruption:b2cov.productivity
-3.1447822
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
2.761880e-01 2.233322e-01
gwb2deg.fixed.0.25 b1cov.disruption
5.433902e-01 4.729572e-01
b2cov.productivity nodematch.field_broad.SHAPE
8.407720e-01 5.468272e-02
nodematch.field_broad.STEM nodematch.country
1.103717e-01 2.471705e-10
b1cov.disruption:b2cov.productivity
1.662105e-03
Joint P-value (lower = worse): 0.01090894
Chain 2
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
0.6176491 0.3359370
gwb2deg.fixed.0.25 b1cov.disruption
0.5077565 1.9178933
b2cov.productivity nodematch.field_broad.SHAPE
-2.2897297 4.7118362
nodematch.field_broad.STEM nodematch.country
2.2802461 -0.8737307
b1cov.disruption:b2cov.productivity
-4.2864780
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
5.368067e-01 7.369184e-01
gwb2deg.fixed.0.25 b1cov.disruption
6.116241e-01 5.512455e-02
b2cov.productivity nodematch.field_broad.SHAPE
2.203699e-02 2.454947e-06
nodematch.field_broad.STEM nodematch.country
2.259310e-02 3.822649e-01
b1cov.disruption:b2cov.productivity
1.815282e-05
Joint P-value (lower = worse): 0.3850826
Chain 3
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
-1.8353121 4.3228744
gwb2deg.fixed.0.25 b1cov.disruption
-0.8371760 -0.0884824
b2cov.productivity nodematch.field_broad.SHAPE
1.3527641 -2.7821481
nodematch.field_broad.STEM nodematch.country
-5.9100044 2.4164155
b1cov.disruption:b2cov.productivity
-0.5564885
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
6.645946e-02 1.540094e-05
gwb2deg.fixed.0.25 b1cov.disruption
4.024936e-01 9.294933e-01
b2cov.productivity nodematch.field_broad.SHAPE
1.761310e-01 5.400039e-03
nodematch.field_broad.STEM nodematch.country
3.420986e-09 1.567416e-02
b1cov.disruption:b2cov.productivity
5.778769e-01
Joint P-value (lower = worse): 0.0057551
Chain 4
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
-1.19593008 -1.37061493
gwb2deg.fixed.0.25 b1cov.disruption
-0.66395880 -0.97212622
b2cov.productivity nodematch.field_broad.SHAPE
3.53590496 -3.14603546
nodematch.field_broad.STEM nodematch.country
-0.00861083 1.16066656
b1cov.disruption:b2cov.productivity
2.45653345
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
0.2317238447 0.1704950267
gwb2deg.fixed.0.25 b1cov.disruption
0.5067166816 0.3309877575
b2cov.productivity nodematch.field_broad.SHAPE
0.0004063809 0.0016549994
nodematch.field_broad.STEM nodematch.country
0.9931296363 0.2457775244
b1cov.disruption:b2cov.productivity
0.0140284740
Joint P-value (lower = worse): 0.1000172
Chain 5
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
-0.8522244 0.0894545
gwb2deg.fixed.0.25 b1cov.disruption
-1.4583848 3.7292765
b2cov.productivity nodematch.field_broad.SHAPE
0.6511978 1.2460571
nodematch.field_broad.STEM nodematch.country
5.0902365 5.9599412
b1cov.disruption:b2cov.productivity
-7.0898812
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
3.940896e-01 9.287207e-01
gwb2deg.fixed.0.25 b1cov.disruption
1.447345e-01 1.920304e-04
b2cov.productivity nodematch.field_broad.SHAPE
5.149188e-01 2.127434e-01
nodematch.field_broad.STEM nodematch.country
3.576173e-07 2.523288e-09
b1cov.disruption:b2cov.productivity
1.342272e-12
Joint P-value (lower = worse): 0.002801371
Chain 6
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
2.0588218 1.4213467
gwb2deg.fixed.0.25 b1cov.disruption
1.1835111 -3.1567584
b2cov.productivity nodematch.field_broad.SHAPE
0.1854236 -0.3016362
nodematch.field_broad.STEM nodematch.country
1.1389416 -0.4868394
b1cov.disruption:b2cov.productivity
1.3907430
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
0.039511309 0.155215999
gwb2deg.fixed.0.25 b1cov.disruption
0.236606662 0.001595334
b2cov.productivity nodematch.field_broad.SHAPE
0.852896834 0.762929446
nodematch.field_broad.STEM nodematch.country
0.254727523 0.626372176
b1cov.disruption:b2cov.productivity
0.164303380
Joint P-value (lower = worse): 0.06858057
Chain 7
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
-1.0546663 -2.7338237
gwb2deg.fixed.0.25 b1cov.disruption
-0.8714338 -0.2720580
b2cov.productivity nodematch.field_broad.SHAPE
2.2708502 -2.5226094
nodematch.field_broad.STEM nodematch.country
-0.8452067 1.7428079
b1cov.disruption:b2cov.productivity
1.7426948
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
0.291577965 0.006260356
gwb2deg.fixed.0.25 b1cov.disruption
0.383517358 0.785577409
b2cov.productivity nodematch.field_broad.SHAPE
0.023156050 0.011648770
nodematch.field_broad.STEM nodematch.country
0.397995424 0.081367174
b1cov.disruption:b2cov.productivity
0.081386948
Joint P-value (lower = worse): 0.0331198
Chain 8
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
-1.6446431 -1.6903388
gwb2deg.fixed.0.25 b1cov.disruption
-1.8346366 1.6174488
b2cov.productivity nodematch.field_broad.SHAPE
3.1551526 -0.5893164
nodematch.field_broad.STEM nodematch.country
2.7023242 0.2752923
b1cov.disruption:b2cov.productivity
10.4749900
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
1.000434e-01 9.096316e-02
gwb2deg.fixed.0.25 b1cov.disruption
6.655955e-02 1.057814e-01
b2cov.productivity nodematch.field_broad.SHAPE
1.604142e-03 5.556490e-01
nodematch.field_broad.STEM nodematch.country
6.885659e-03 7.830917e-01
b1cov.disruption:b2cov.productivity
1.125495e-25
Joint P-value (lower = worse): 5.59447e-09
Chain 9
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
-1.11221542 1.68341731
gwb2deg.fixed.0.25 b1cov.disruption
-0.03171701 -1.22821678
b2cov.productivity nodematch.field_broad.SHAPE
2.66349533 -0.61010950
nodematch.field_broad.STEM nodematch.country
-1.81587979 -2.83278285
b1cov.disruption:b2cov.productivity
-2.39552426
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
0.266045541 0.092294335
gwb2deg.fixed.0.25 b1cov.disruption
0.974697732 0.219365600
b2cov.productivity nodematch.field_broad.SHAPE
0.007733349 0.541789272
nodematch.field_broad.STEM nodematch.country
0.069388814 0.004614472
b1cov.disruption:b2cov.productivity
0.016596616
Joint P-value (lower = worse): 0.1177789
Chain 10
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
-1.38516164 1.03344851
gwb2deg.fixed.0.25 b1cov.disruption
-2.46519276 -0.78416088
b2cov.productivity nodematch.field_broad.SHAPE
0.63959208 -0.06279892
nodematch.field_broad.STEM nodematch.country
1.71558227 -2.43480486
b1cov.disruption:b2cov.productivity
4.17780822
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
1.660030e-01 3.013941e-01
gwb2deg.fixed.0.25 b1cov.disruption
1.369396e-02 4.329457e-01
b2cov.productivity nodematch.field_broad.SHAPE
5.224378e-01 9.499266e-01
nodematch.field_broad.STEM nodematch.country
8.623853e-02 1.489982e-02
b1cov.disruption:b2cov.productivity
2.943317e-05
Joint P-value (lower = worse): 0.003450341
Chain 11
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
-2.3760814 3.0663764
gwb2deg.fixed.0.25 b1cov.disruption
-2.8099727 -3.4027425
b2cov.productivity nodematch.field_broad.SHAPE
0.8168717 4.2903879
nodematch.field_broad.STEM nodematch.country
-1.3369505 3.6104786
b1cov.disruption:b2cov.productivity
-5.7441819
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
1.749760e-02 2.166704e-03
gwb2deg.fixed.0.25 b1cov.disruption
4.954570e-03 6.671311e-04
b2cov.productivity nodematch.field_broad.SHAPE
4.140017e-01 1.783613e-05
nodematch.field_broad.STEM nodematch.country
1.812388e-01 3.056325e-04
b1cov.disruption:b2cov.productivity
9.236643e-09
Joint P-value (lower = worse): 0.0003736895
Chain 12
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
-0.5841887 -1.1962406
gwb2deg.fixed.0.25 b1cov.disruption
-0.5346914 -1.6584206
b2cov.productivity nodematch.field_broad.SHAPE
-0.6019855 -3.1572403
nodematch.field_broad.STEM nodematch.country
-0.3356684 -2.2209444
b1cov.disruption:b2cov.productivity
0.8905005
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
0.55909338 0.23160269
gwb2deg.fixed.0.25 b1cov.disruption
0.59286329 0.09723260
b2cov.productivity nodematch.field_broad.SHAPE
0.54718378 0.00159270
nodematch.field_broad.STEM nodematch.country
0.73712091 0.02635473
b1cov.disruption:b2cov.productivity
0.37319722
Joint P-value (lower = worse): 0.1324312
Chain 13
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
-0.9648019 0.1849311
gwb2deg.fixed.0.25 b1cov.disruption
-0.2017320 -0.4905972
b2cov.productivity nodematch.field_broad.SHAPE
2.2229655 -6.5008081
nodematch.field_broad.STEM nodematch.country
-3.2891326 -2.7704883
b1cov.disruption:b2cov.productivity
-5.3446596
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
3.346440e-01 8.532831e-01
gwb2deg.fixed.0.25 b1cov.disruption
8.401263e-01 6.237114e-01
b2cov.productivity nodematch.field_broad.SHAPE
2.621813e-02 7.988970e-11
nodematch.field_broad.STEM nodematch.country
1.004967e-03 5.597231e-03
b1cov.disruption:b2cov.productivity
9.058711e-08
Joint P-value (lower = worse): 6.643703e-05
Chain 14
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
-1.2852513 0.7976323
gwb2deg.fixed.0.25 b1cov.disruption
0.1942386 -0.3139755
b2cov.productivity nodematch.field_broad.SHAPE
-1.1409346 -1.1679390
nodematch.field_broad.STEM nodematch.country
-1.4361457 0.7344049
b1cov.disruption:b2cov.productivity
2.3299071
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
0.19870448 0.42508390
gwb2deg.fixed.0.25 b1cov.disruption
0.84598908 0.75353962
b2cov.productivity nodematch.field_broad.SHAPE
0.25389715 0.24283136
nodematch.field_broad.STEM nodematch.country
0.15096087 0.46270198
b1cov.disruption:b2cov.productivity
0.01981106
Joint P-value (lower = worse): 0.008978321
Chain 15
Fraction in 1st window = 0.1
Fraction in 2nd window = 0.5
edges gwb1deg.fixed.0.25
-0.7056351 0.2137390
gwb2deg.fixed.0.25 b1cov.disruption
-0.3473258 0.6798398
b2cov.productivity nodematch.field_broad.SHAPE
1.5079380 -1.2780596
nodematch.field_broad.STEM nodematch.country
-5.5733713 2.9645932
b1cov.disruption:b2cov.productivity
4.4896129
Individual P-values (lower = worse):
edges gwb1deg.fixed.0.25
4.804151e-01 8.307506e-01
gwb2deg.fixed.0.25 b1cov.disruption
7.283465e-01 4.966059e-01
b2cov.productivity nodematch.field_broad.SHAPE
1.315704e-01 2.012284e-01
nodematch.field_broad.STEM nodematch.country
2.498562e-08 3.030834e-03
b1cov.disruption:b2cov.productivity
7.135272e-06
Joint P-value (lower = worse): 5.31663e-07
Note: MCMC diagnostics shown here are from the last round of
simulation, prior to computation of final parameter estimates.
Because the final estimates are refinements of those used for this
simulation run, these diagnostics may understate model performance.
To directly assess the performance of the final model on in-model
statistics, please use the GOF command: gof(ergmFitObject,
GOF=~model).Interpretación de Resultados del ERGM
Los resultados del modelo permiten evaluar nuestras hipótesis sobre la estructura del campo científico:
- H1: Estructura Base y Centralización
Los resultados confirman parcialmente esta hipótesis. El coeficiente negativo y significativo de edges (β = -9.11, p < 0.001) indica una baja densidad base en la red, consistente con la predicción de Gondal (2011) sobre la incertidumbre estructural en campos emergentes. Sin embargo, la centralización muestra un patrón más complejo que el esperado: Tanto papers (gwb1deg: β = 3.04, p < 0.001) como autores (gwb2deg: β = 8.44, p < 0.001) muestran tendencias significativas a la centralización. La magnitud es notablemente mayor para autores, sugiriendo un rol más prominente en la estructuración del campo.
- H2: Asimetría Disciplinar
La hipótesis sobre asimetría disciplinar se confirma con matices importantes. Los resultados muestran patrones direccionales claros:
- STEM muestra homofilia positiva significativa (β = 0.89, p < 0.001)
- SHAPE exhibe homofilia negativa significativa (β = -0.82, p < 0.001)
Esto refina la tesis de Friedkin (1998) sobre “consensos superpuestos”, sugiriendo que la integración del conocimiento es direccional, con STEM mostrando cohesión interna mientras SHAPE tiende a vínculos interdisciplinares.
- H3: Dinámica Centro-Periferia y Disrupción
Contrario a lo predicho por Li et al. (2024), no encontramos evidencia de una relación significativa entre:
- Disrupción y formación de vínculos (β = 0.047, p = 0.508)
- Productividad y formación de vínculos (β = -0.095, p = 0.616)
- Su interacción (β = -0.001, p = 0.981)
Esto sugiere que la relación entre posición estructural y capacidad disruptiva puede ser más compleja que lo inicialmente teorizado.
- H4: Evolución Temporal y Campos Emergentes
La evidencia sobre la evolución temporal es mixta. La significativa centralización en ambos modos (gwb1deg y gwb2deg) sugiere que el campo ha establecido mecanismos de legitimación, pero los patrones de interacción disciplinar indican una estructura más compleja que la simple centralización-periferia propuesta por Gondal (2011).
- H5: Integración Disciplinar Asimétrica
Esta hipótesis recibe el apoyo más fuerte. Los patrones de homofilia muestran una clara diferenciación:
- STEM muestra fuerte cohesión interna (β = 0.89, p < 0.001)
- SHAPE muestra tendencia a vínculos externos (β = -0.82, p < 0.001)
- La homofilia geográfica no es significativa (β = -0.08, p = 0.613)
Implicaciones Teóricas
Refinamiento de la Teoría de Campos Emergentes: Los resultados sugieren que la teoría de Gondal (2011) necesita ser refinada para incorporar:
- Centralización multi-modal con efectos asimétricos entre autores y papers
- Patrones disciplinares diferenciados en la formación de vínculos
- Roles estructurales específicos por campo científico
Reconsideración de la Relación Disrupción-Estructura: La ausencia de efectos significativos para disrupción y productividad sugiere que el marco de Li et al. (2024) y Wu et al. (2019) necesita ser reconsiderado en el contexto de redes modo-2, donde la relación entre posición estructural e innovación puede operar de manera diferente.
Estructuras Híbridas y Tensiones Productivas: El patrón observado sugiere que los campos emergentes desarrollan estructuras híbridas caracterizadas por:
- Centralización significativa pero asimétrica entre modos
- Integración disciplinar diferenciada (STEM cohesivo, SHAPE puente)
- Patrones de colaboración independientes de la geografía